Diabetesforschung Innovatives Datenmanagement gegen Diabetes
 Intelligent vernetzte Informationen statt unübersichtliche Datensilos.
© iStock/metamorworks
Intelligent vernetzte Informationen statt unübersichtliche Datensilos.
© iStock/metamorworks
In Deutschland gibt es beim Datenmanagement in der Forschung immensen Nachholbedarf. Riesige Datenmengen sind das eine – deren Organisation und Verwertbarkeit das andere. Schuld daran sind gleich mehrere Faktoren: Medizinische Daten sind im Normalfall heterogen. Sie bestehen z.B. aus Röntgenbildern, Blutbildern und Blutzuckerwerten, die in pdf-, jpeg- oder sonstigen Dateiformaten vorliegen. Diese Heterogenität stellt eine große Hürde für effizientes Arbeiten dar. Außerdem ist der Datenschatz oft auf verschiedene Forschungsstandorte verteilt – und somit völlig dezentral. Hinzu kommt, dass die Menge der Informationen schnell und kontinuierlich wächst. Was bleibt, ist eine Reihe von unvernetzten Datensilos.
Semantische Abfragen, intuitive Darstellung im Graph
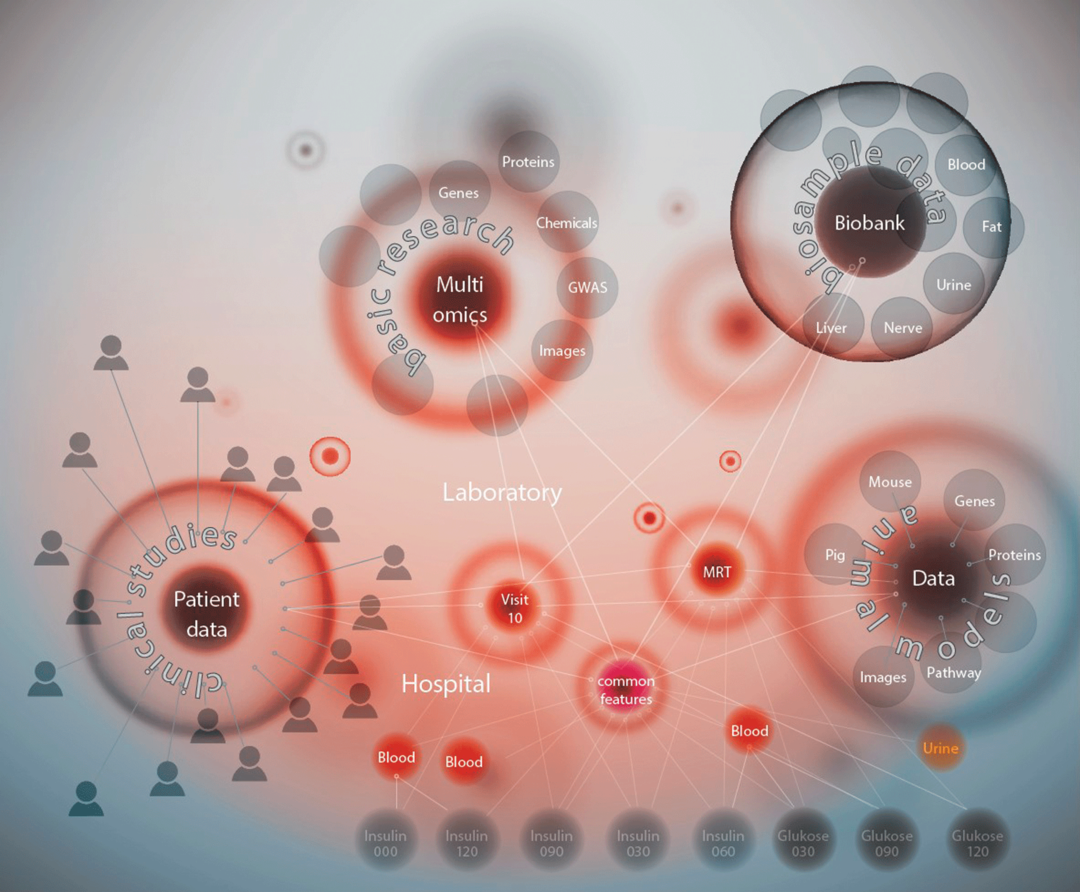
Selbst wenn alle Dokumente sauber und zentral vorliegen, besteht noch ein Problem: Große Datenmengen allein schaffen noch kein Wissen. Um aus der heterogenen Masse neue Erkenntnisse und somit einen Mehrwert herauszuziehen, muss man einen genauen Blick auf die Beziehungen zwischen den unterschiedlichen Daten werfen. Graphtechnologie und sogenannte Knowledge Graphen tun genau das – sie stellen Beziehungen zwischen Daten her und helfen dabei, neue Erkenntnisse zu gewinnen.
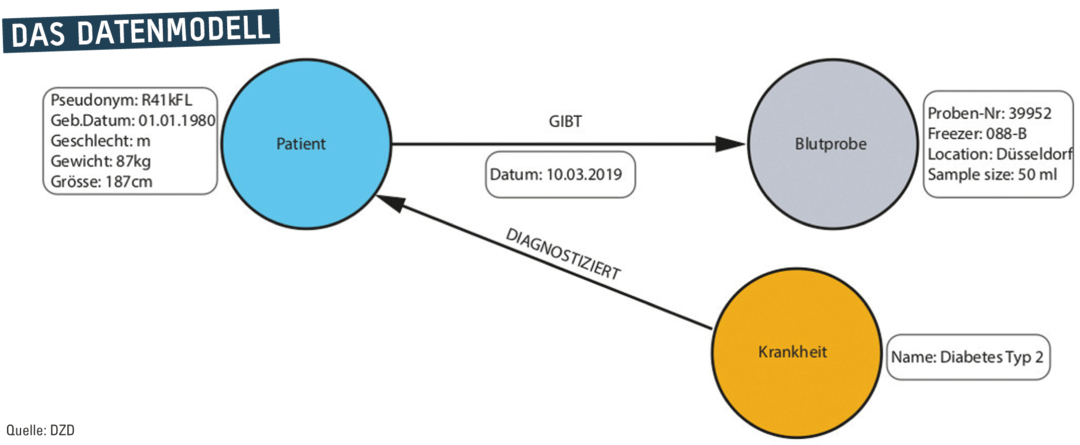
Um die Funktionsweise von Graphdatenbanken zu verstehen, muss man sich zuerst mit dem Datenmodell an sich beschäftigen. Daten werden in einem Graphen als Kreise (Knoten) dargestellt, die über Linien (Kanten) miteinander verbunden sind. Beiden können qualitative oder quantitative Eigenschaften zugeordnet werden. Neue Informationen fügen Data Scientists beliebig über das Knoten-Kanten-Prinzip hinzu. So können auch große Netzwerke aus Beziehungen und Zusammenhängen übersichtlich dargestellt werden. Auch der komplexe Organismus Mensch lässt sich so realitätsnah abbilden – mit allen Genen, Proteinen und Faktoren, die für Diabetes eine Rolle spielen.
Graphdatenbanken sind eine echte Alternative zu relationalen Datenbanken. Denn diese speichern Informationen in Zeilen und Spalten. Entsprechend intensiv ist die Rechenleistung, wenn es darum geht, die Verbindungen zwischen den Daten abzufragen. Dadurch steigen die Kosten und es kommt im schlimmsten Fall zum Systemausfall. In Graphdatenbanken hingegen bleibt die Performance unabhängig von Größe und Komplexität der Daten konstant. Abfragen lassen sich in Millisekunden, also in Echtzeit, durchführen.
In Graphdatenbanken lassen sich zudem komplexe Prozesse wie in einer Mind-Map anschaulich visualisieren. Es entsteht ein semantischer Kontext, um investigativ zu forschen. Damit gewinnen Forschungseinrichtungen nicht nur einen Wissensvorsprung. Sie erhalten auch eine smarte Basistechnologie für Künstliche Intelligenz (KI). Denn Graphen stellen prädikative Parameter zur Verfügung und bereiten so dem Einsatz von Machine Learning und KI den Weg.
Um unvernetzte Datensilos in den Griff zu bekommen, baute das DZD 2017 ein standortübergreifendes Daten- und Knowledge Management auf: DZDconnect.
Paradebeispiel Diabetesforschung: das DZDconnect
Dieses System wird von 450 Healthcare und Medical Professionals genutzt. Es basiert auf der Graphdatenbank Neo4j und erstellt Verknüpfungen zwischen Metadaten aus Grundlagenforschung und klinischen Studien. Neo4j liegt hierbei als Layer über den relationalen Datenbanken und verknüpft die Systeme und Datensilos der Gesundheitszentren. Über das Visualisierungstool Neo4j Bloom sind Fragen in natürlicher Sprache möglich: z.B. wie viele Blutproben von männlichen Patienten unter 69 Jahren haben wir?
NLP für einen ganzheitlichen Forschungsansatz
Mithilfe von Natural Language Processing (NLP) gelang es, über 30 Mio. Veröffentlichungen aus der Literaturdatenbank PubMed einzulesen und automatisch zu annotieren. Derzeit dauert es circa 1,5 Sekunden, einen Abstract zu analysieren. Das klingt zwar schnell, tatsächlich hätte es aber rund eineinhalb Jahre gedauert, um alle 30 Mio. Veröffentlichungen zu analysieren. Der Ansatz von NLP und Graphdatenbank des DZD läuft daher parallel und automatisiert im Hintergrund ab. Dank ausgeklügelter Algorithmen wird eine semantische Analyse der Texte durchgeführt. Hierbei erkennt die Technologie relevante Entitäten und verknüpft sie mit internen Informationen in der Datenbank. Diese Art des „Lesens“ ist beachtlich schneller als das menschliche Auge. Ein ganzheitlicher Ansatz ist das Ziel der Forschung. Dank NLP und Graphtechnologie können Forscher auch andere wichtige Volkskrankheiten mit einbeziehen. So erreichen sie das Ziel, medizinische Fragen aus unterschiedlichen Blickwinkeln zu betrachten und indikationsübergreifend zu arbeiten.
In der nächsten Projektphase ergänzten Wissenschaftler die humanen Daten aus der klinischen Forschung mit hochstandardisierten Daten aus Tiermodellen, z.B. aus Mäusen. Das dient dazu, Gemeinsamkeiten zu erkennen und Rückschlüsse zu ziehen. Die (Meta-)Daten wurden um disziplinübergreifende Forschungsdaten aus öffentlichen Quellen erweitert – dazu gehören Daten zu anderen relevanten Krankheiten. Diese Verknüpfung erlaubt es, Schlussfolgerungen für das humane System zu ziehen und Ähnlichkeiten bei einzelnen Genen und Stoffwechselvorgängen zu untersuchen. Hierbei suchen Wissenschaftler Antworten auf Fragestellungen wie: Welcher Diabetes-Typ lässt sich auf welche Gene zurückführen? Wie wirken sich externe Faktoren aus?
Bei der Beantwortung dieser Fragen helfen integrierte Graph-Algorithmen wie Community-Detection. Dieser Algorithmus erkennt auf Basis vorab definierter Parameter Patienten-Cluster. Die DZD-Mitarbeitenden können diese dadurch genauer untersuchen. Dabei werden die jeweiligen Charakteristiken der Diabetes-Subtypen präzisiert und Gemeinsamkeiten (z.B. Größe, Gewicht, Medikation oder Gendefekt) bestimmt. Das DZD forscht derzeit an Subtypen des Typ-2-Diabetes (Stichwort: Precision Medicine). Die Graph Data Science Library von Neo4j übernimmt dabei eine wichtige Rolle: Mithilfe der integrierten Algorithmen können Wissenschaftler das Datenset unterteilen.
Insgesamt umfasst der DZD Knowledge Graph 1,8 Mrd. Knoten und 4,9 Mrd. Kanten. Im Graphen lassen sich alle relevanten Daten ganzheitlich abbilden.